Vergleichende Genomik
Der evolutionäre Ursprung von Nesselzellen im Stechenden Schwarzen Schwamm Haliclona cnidata
Institution: Justus-Liebig-Universität Gießen
Sie sind unauffällig und doch überaus erfolgreich – Meeresschwämme sind evolutionäre Meister der Verteidigung und verfügen in der Regel über ein hohes Maß an Bioaktivität, um Räuber und Konkurrenten abzuschrecken. Beim Stechenden Schwarzen Schwamm (Haliclona cnidata) werden diese Mechanismen durch „zelluläre Waffen“ in Form von funktionalen Nesselzellen ergänzt (siehe Abbildung 1), für die sonst nur Nesseltiere wie Quallen oder Korallen bekannt sind.
Woher diese Nesselzellen stammen, ist noch weitgehend unbekannt. Der Schwamm könnte die Zellen durch Kleptocnidismus aufnehmen (wörtlich übersetzt das Klauen von Nesselzellen). Dabei werden Nesselzellen oder deren Vorläuferstadien von anderen Nesseltieren wie Korallen aus der Umgebung aufgenommen und eingelagert. Vielleicht lebt er aber auch in einer Symbiose mit Nesseltieren, die in seinem Gewebe Schutz finden und im Gegenzug dazu Nesselzellen bilden und ihm zur Verfügung stellen. Oder er hat sogar Gene der Nesseltiere in sein eigenes Genom übernommen und kann diese Zellen nun selbstständig bilden.
Dem Ursprung der Nesselzellen im Stechenden Schwarzen Schwamm wollen wir in unserem Forschungsprojekt „Der evolutionäre Ursprung von Nesselzellen im Stechenden Schwarzen Schwamm Haliclona cnidata“ im Fachgebiet Tierökologie und Spezielle Zoologie an der Justus-Liebig-Universität Gießen (JLU) mit experimenteller Laborforschung und Genomanalysen auf den Grund gehen.
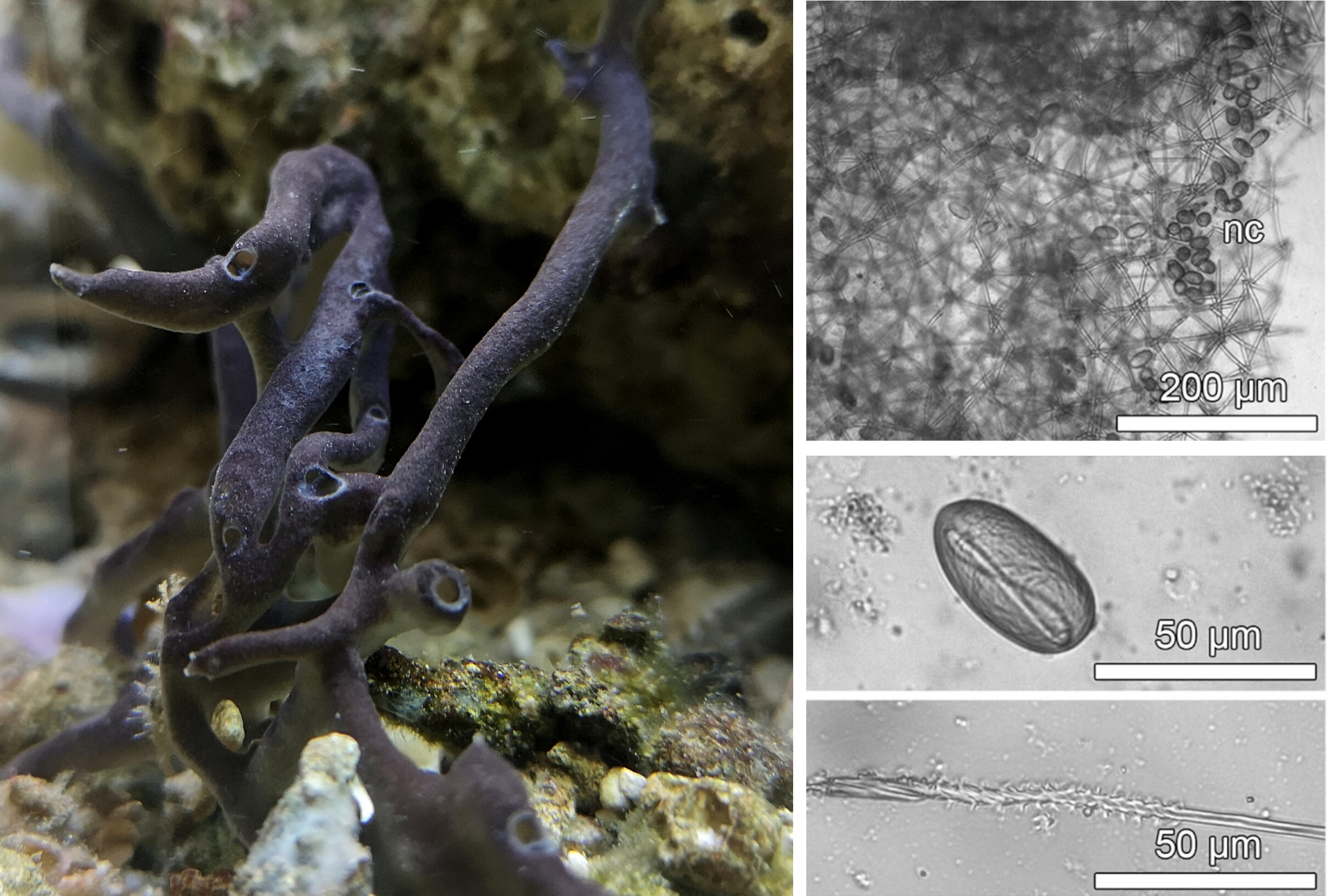
Abbildung 1: Schwämme kommen in marinen Lebensräumen als wichtige Bestandteile des benthischen Ökosystems vor. Der Stechende Schwarze Schwamm (Haliclona cnidata) ist leicht zu übersehen und wurde bisher nur in tropischen Meerwasseraquarien gefunden (links). Zu seiner Verteidigung hat er, wie auch Nesseltiere, Nesselzellen (nc) in der äußeren Schicht seines Gewebes eingelagert, die bei Gefahr ausgelöst werden und ein hochwirksames Gift in den Angreifer injizieren können (rechts). Quelle: Schellenberg et al. 2019
Expertise, Methoden
- Struktur und Funktion von Metaorganismen
- Modellorganismen
- Symbiose
- Analyse mikrobieller Gemeinschaften
- Morphometrische Untersuchungen
- Experimentelle Meeresbiologie
- Simulationen des globalen Wandels
- Ökophysiologie
genomdaten
Im Rahmen des Projekts wird das Genoms des Schwamms sequenziert. Bei der bioinformatischen Analyse werden dabei neben dem Schwamm auch eine große Zahl mikrobieller Symbionten identifiziert. Als sessile Filtrierer sind Schwämme ständig Mikroorganismen aus der Umwelt ausgesetzt, die sowohl vorteilhaft als auch schädlich für den Schwamm sein können. Haliclona cnidata zählt zudem zu den „high microbial abundance“ (HMA) Schwämmen und beherbergt eine komplexe, schwammspezifische und evolutionär angepasste mikrobielle Gemeinschaft (Abbildung 2), die gemeinsam mit dem Schwamm eine funktionelle Einheit bildet – den Schwammholobionten. In den nächsten Schritten ist geplant den Schwamm isoliert von seinen Symbionten zu analysieren, um so einen bessere Datengrundlage für die bioinformatischen Analysen zu schaffen. In experimentellen Laboruntersuchungen wird die Bildung der Nesselzellen unter kontrollierten Bedingungen genauer untersucht.
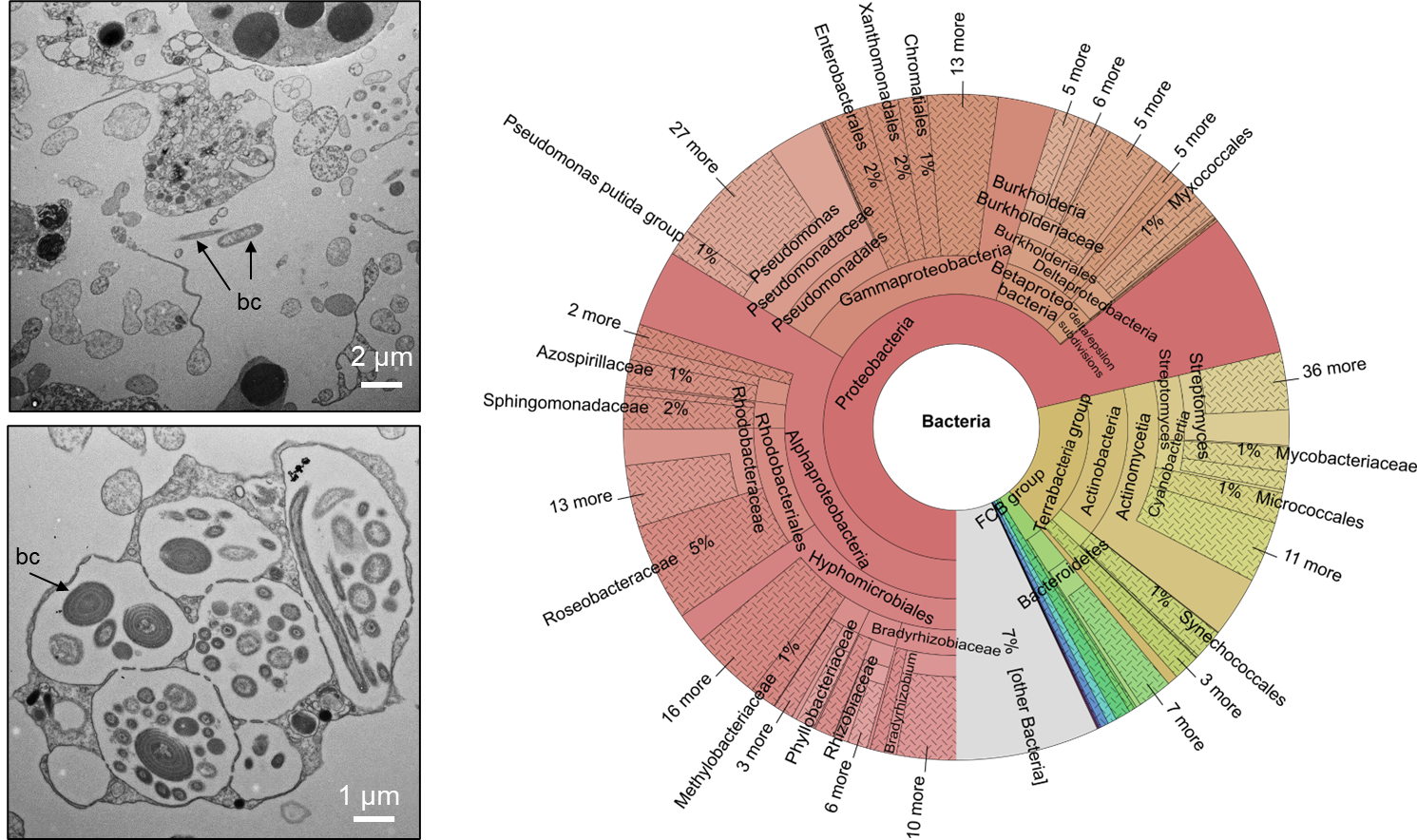
Abbildung 2: Bakterien (bc) leben im Stechenden Schwarzen Schwamm gelegentlich im Mesohyl (links oben) und häufig in spezialisierten Kompartimenten, den sogenannten Bakteriocyten, wie hier in einer transmissionselektronenmikroskopischen (TEM) Aufnahme zu sehen ist (links unten). Die komplexe bakterielle Gemeinschaft wurde in einer ersten Studie basierend auf der Genomsequenzierung des Holobionten analysiert (rechts). Quellen: Schellenberg et al. 2020, Pfeiffer 2021.
Kooperationsprojekte
Der Schwamm wurde im Rahmen der Doktorarbeit von Johannes Schellenberg im Institut für Tierökologie und Spezielle Zoologie an der Justus-Liebig-Universität Gießen erstmalig untersucht und beschrieben (Schellenberg et al., 2018, 2019, 2020). Erste Genomanalysen fanden im Rahmen der Masterarbeit von Yvonne Pfeifer in Kooperation mit Jörn Kalinowski und Daniel Wibberg vom CeBiTec in Bielefeld statt. Dabei wurde Schwammgewebe des Schwammholobionten verwendet, um das Genom zusammenzusetzen und die Symbiontengemeinschaft des Schwamms zu charakterisieren.
Die Untersuchung des Schwamms ist ein Kollaborationsprojekt, das in der „Ocean2100“ Aquarienanlage der Uni Gießen stattfindet. Die „Ocean2100“ Anlage ist Teil einer vom DAAD (Deutscher Akademischer Austauschdienst) geförderten Exzellenzinitiative des CEMarin (Center of Excellence in Marine Sciences, in der globale Veränderungen im Meer simuliert werden und Steinkorallen und andere in Korallenriffen lebende Organismen verschiedenen Veränderungsszenarien ausgesetzt werden können.
Ausgewählte Publikationen
- Schellenberg, J., Busse, H. J., Hardt, M., Schubert, P., Wilke, T., Käampfer, P., & Glaeser, S. P. (2018). Proposal of Litorimonas haliclonae sp. Nov., isolated from a marine sponge of the genus Haliclona. International Journal of Systematic and Evolutionary Microbiology, 68(3), 835–843. https://doi.org/10.1099/ijsem.0.002592
- Schellenberg, J., Reichert, J., Hardt, M., Klingelhöfer, I., Morlock, G., Schubert, P., Bižić, M., Grossart, H. P., Kämpfer, P., Wilke, T., & Glaeser, S. P. (2020). The Bacterial Microbiome of the Long-Term Aquarium Cultured High-Microbial Abundance Sponge Haliclona cnidata – Sustained Bioactivity Despite Community Shifts Under Detrimental Conditions. Frontiers in Marine Science, 7(May). https://doi.org/10.3389/fmars.2020.00266
- Schellenberg, J., Reichert, J., Hardt, M., Schmidtberg, H., Kämpfer, P., Glaeser, S. P., Schubert, P., & Wilke, T. (2019). The precursor hypothesis of sponge kleptocnidism: Development of nematocysts in Haliclona cnidata sp. nov. (porifera, demospongiae, haplosclerida). Frontiers in Marine Science, 5(JAN), 1–14. https://doi.org/10.3389/fmars.2018.00509