TBG Laborzentrum
Dr. Carola Greve, Leonie Schardt, Damian Baranski und Alexander Ben Hamadou
Gemeinsam mit der Serviceeinheit Bioinfomatik hat das TBG-Labor ein Datenmanagementsystem aufgebaut, das die Roh- und Metadaten von >3000 Sequenzierläufen (Stand 08/2021) organisiert und speichert, um sie für alle PIs am TBG verfügbar zu machen und die Einreichung der Daten vorzubereiten.



Wir sind gut ausgestattet, um die Herausforderungen bei der Isolierung von hmwDNA aus Säugetieren, Mollusken und Insekten bis hin zu Pflanzen, Flechten und Pilzen zu meistern. Als PacBio-Beta-Tester sequenzieren wir ganze de novo Genome von winzigen Organismen (1-2 mm) mit einer Inputmenge von nur 5 ng gDNA. Mit unserer Geräteausstattung können wir alle notwendigen Qualitätskontrollen durchführen, einschließlich der Abschätzung der Genomgröße, der Pulsfeldelektrophorese und der DNA Fragmentierung, um NGS Bibliotheken herzustellen, die wir bei verschiedenen Kooperationspartnern (BGI, Novogene, Radboud University Medical Center, SciLife) sequenzieren. Ein Teil der Sequenzierung wird intern mit dem Oxford Nanopore (etwa 60 Genome) oder der Illumina Plattform iSeq 100 für Qualitätstests und kleine Projekte durchgeführt.
Ausstattung
- Pulsfeld Elektrophorese (Analytik Jena)
- 2200 TapeStation (Agilent Technologies)
- Qubit 2.0 (Life Technologies)
- BluePippin (Sage Science)
- MinION (Oxford Nanopore)
- Illumina Next Generation Sequencing (NGS) platform iSeq 100
- Bioruptor ® Pico (Diagenode)
- CytoFLEX Flow Cytometer (Beckman Coulter)
- Femtopulse (Agilent)
- Bioanalyzer (Agilent)
- Plate reader (M2, Molecular devices)
- qPCR-Maschine (CFX Opus 96, Bio-Rad)
- 2x Thermocycler (Mastercycler X50t, Eppendorf)
- Pipettierroboter
M220 Focused-ultrasonicator
SEQUENZIERTE GENOME
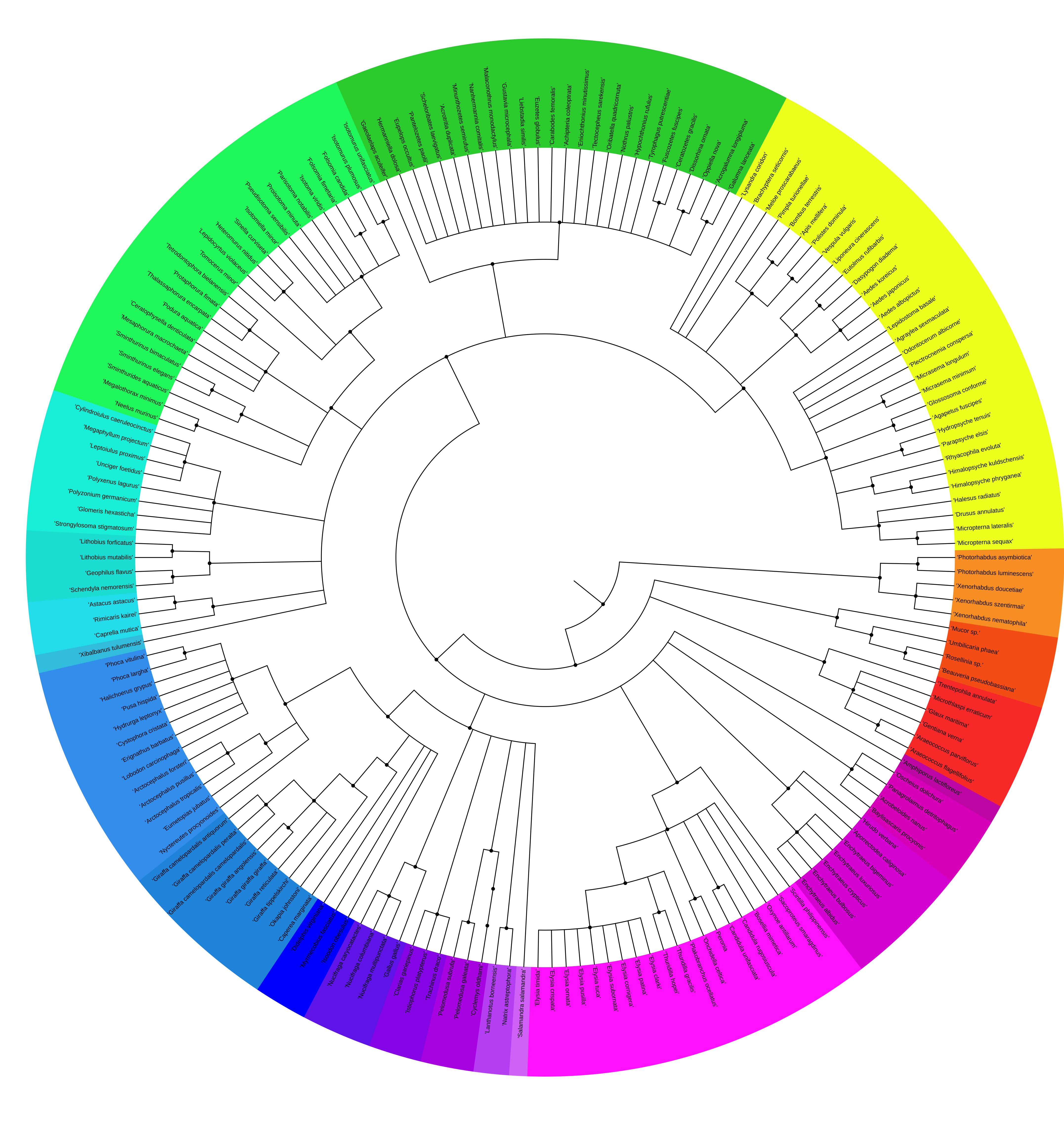
Sequenzierte Genome“: Übersicht der von TBG de novo assemblierten Arten: 351 Arten aus 11 Stämmen.
TEAM TBG Laborzentrum

Dr. Carola Greve (Labor- und Projektmanagerin)
carola.greve@senckenberg.de
Leonie Schardt (Labormanagerin FUG „Funktionale Umweltgenomik“)
leonie.schardt@senckenberg.de
Damian Baranski (Lab Manager Robotics „Funktionale Umweltgenomik“ und „Genomisches Biomonitoring“)
damian.baranski@senckenberg.de
Alexander Ben Hamadou (Technischer Assistent)
alexander-ben.hamadou@senckenberg.de
Charlotte Gerheim (Technische Assistentin)
charlotte.gerheim@senckenberg.de



