Genomische Biomonitoring
eDNA Chips
Institution: Naturschutzgenetik, Senckenberg Gesellschaft für Naturforschung (Gelnhausen)
Ziel des Projektes ist die Entwicklung und der Anwendungstransfer von genomischen Mik- rofluid-Chips zur Erkennung von Artengemeinschaften und Populationsstrukturen anhand von Umweltproben. Dazu wurde eine Standardmethode zur Entwicklung und Testung von Mikrofluid-SNP-Detektionschips für das Erkennen von Populationsstrukturen, Hybridisierungsereignissen und Verwandtschaften innerhalb von Populationen mehrerer bedrohter Arten entwickelt, die in der EU Fauna-Flora-Habitat Richtlinie prioritär gelistet werden. Diese Detektionschips befinden sich bereits in der praktischen Anwendung (z.B. Wildkatze, Wolf, Wisent). Der schnelle Transfer in die Biomonitoring- und Artenschutzpraxis ist über das Zentrum für Wildtiergenetik (SGN Gelnhausen) gewährleistet, welches eine Schnittstelle zwischen Forschung und Anwendung bildet.
- Genotypenbestimmung von forensischen und Umweltproben auf der Basis von Mikrosatelliten und SNPs
- Mikrofluidische SNP-Genotypisierung (Fluidigm)
- Molekulare Populationsgenetik
- Naturschutzgenetik/-genomik
- Wildtierbiologie






Die Wasserprobenahme erfolgt über Filter mit unterschiedlichen Porengrößen, hier über einen Glasfaserfilter mit 2 µm Porengröße. Ist die maximale Filtrationskapazität erreicht, werden die Filter aus dem Filtergehäuse entnommen und stehen für nachfolgende eDNA-Aufreinigung und molekularbiologischen Analysen bereit.
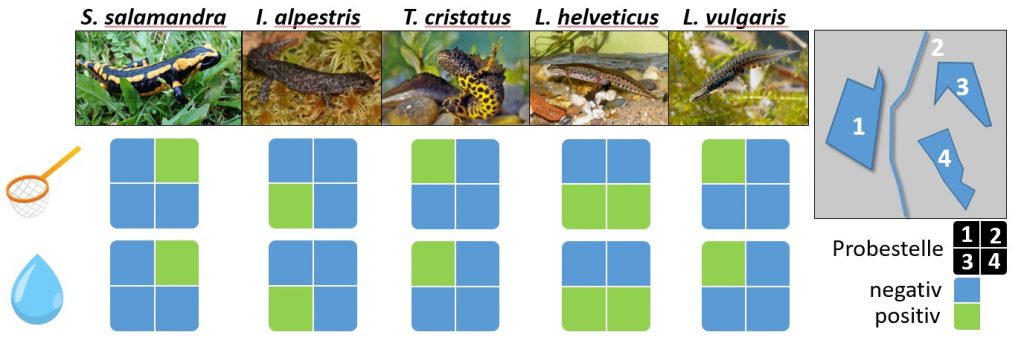
Detektion heimischer Molche: Klassisches Monitoring vs. eDNA Chip an vier Tümpeln

Die Mikrofluid-Technologie erlaubt die parallele Analyse von Tausenden Real-Time-PCR-Reaktionen auf einem Chip. Das spart Zeit und Kosten und minimiert den Verbrauch von Probenmaterial.
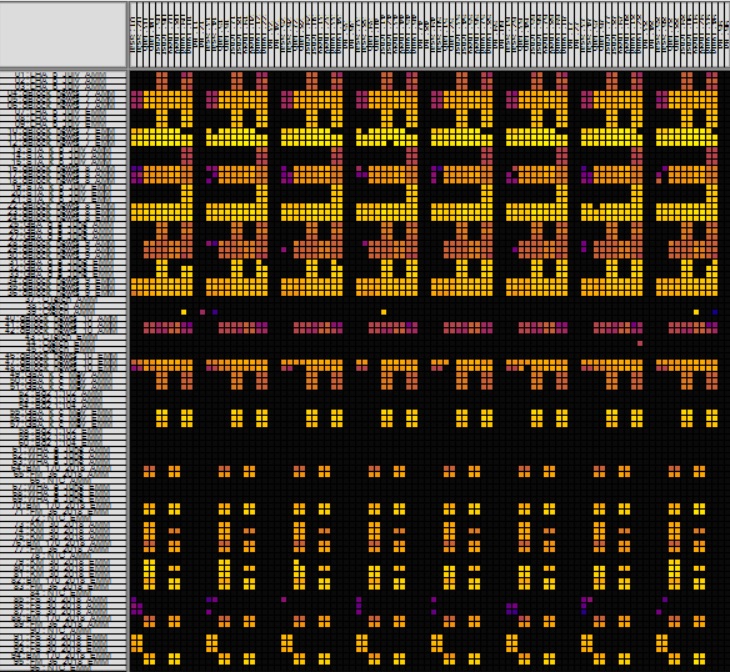
Heatmap-Ansicht der Rohdaten eines Runs mit dem Biomark HD Mikrofluidsystem (FluidigmTM) von eDNA-Proben einheimischer Molcharten
PUBLIKATIONEN
von Thaden A, Nowak C, Tiesmeyer A, et al. (2020). Applying genomic data in wildlife monitoring: Development guidelines for genotyping degraded samples with reduced single nucleotide polymorphism panels. Molecular Ecology Resources 20(3), 662-680.
Harmoinen J, von Thaden A, Aspi J, et al. (2021). Reliable wolf-dog hybrid detection in Europe using a reduced SNP panel developed for non-invasively collected samples. BMC Genomics 22, 473.