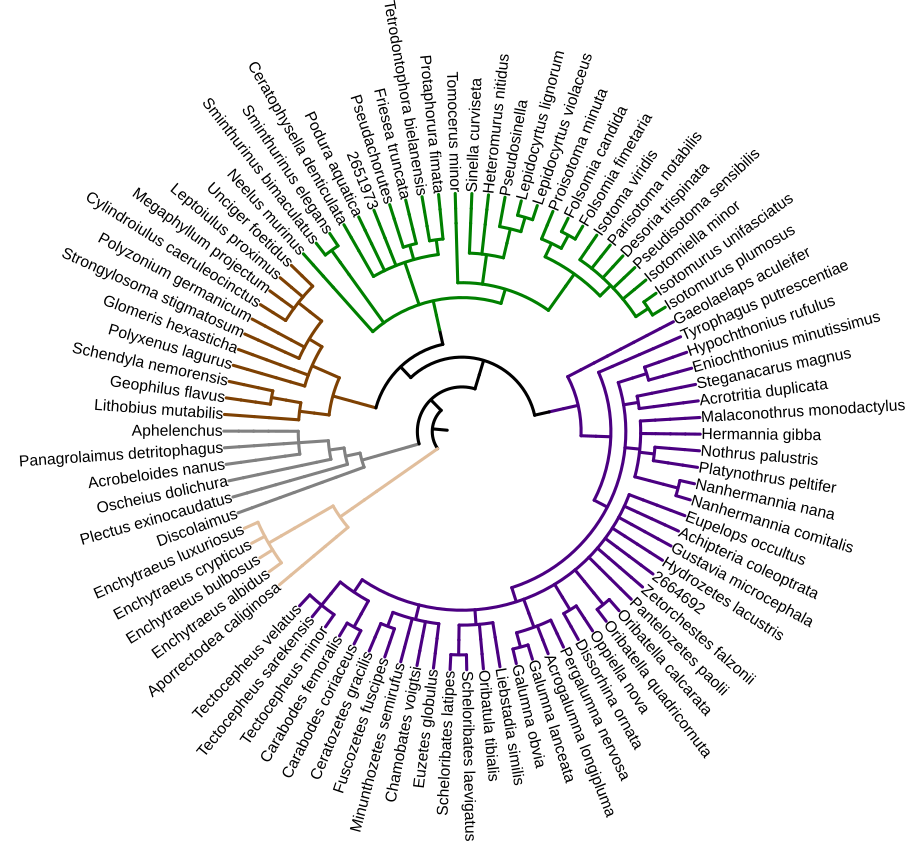Funktionale Umweltgenomik
Metagenomisches Monitoring von Bodengemeinschaften (MetaInvert)
Institution: Senckenberg Gesellschaft für Naturforschung
Direkt unter unseren Füßen gibt es eine enorme Vielfalt an wirbellosen Tieren: Ein einziger Quadratmeter Boden in Deutschland kann hunderte von Arten enthalten. Ein großer Teil dieser Vielfalt ist unbekannt: Man schätzt, dass auf jede bekannte Art etwa zwanzig noch unbeschriebene kommen. Diese wirbellosen Tiere leisten einen wichtigen Beitrag zu Ökosystemdienstleistungen, die für die Menschheit essentiell sind, wie z.B. Krankheitsbekämpfung, Nährstoffkreislauf, Bodenbildung oder sogar Primärproduktion.
Es besteht die ernste Sorge, dass diese Vielfalt schnell verloren geht. Da sich der Verlust der biologischen Vielfalt im Boden wahrscheinlich auf die Ökosystemdienstleistungen des Bodens auswirkt, fordern die nationalen und europäischen Behörden, die biologische Vielfalt im Boden zu verstehen und zu überwachen. Ein umfassendes Monitoring der biologischen Vielfalt im Boden ist jedoch noch nicht möglich, da Methoden fehlen, die es erlauben würden, die Zusammensetzung der Wirbellosengemeinschaften, die Biomasse und die Funktionsvielfalt in großem Maßstab zu beschreiben (siehe Abbildung 1).
Im Projekt MetaInvert entwickeln wir die notwendigen genomischen Ressourcen, Labor- und bioinformatischen Werkzeuge für ein Metagenomik-basiertes Monitoring von wirbellosen Bodengemeinschaften. Zusätzlich ist es unser Ziel den Standardisierungsprozess der relevanten Ansätze durch ISO zu starten.

Abb. 1. Typische Probe von Mikroarthropoden im Boden, die hunderte von Individuen aus wahrscheinlich Dutzenden von Arten enthält. Die Identifizierung solcher Proben durch Mikroskopie erfordert hochqualifizierte Spezialisten und ist zeitaufwändig.
TBG Projekt-Team
Projekt PIs
Miklos Bálint
Ricarda Lehmitz
Projektbeteiligte
Ina Schaefer
Andreas Kauk
Anna Küchler
Karin Hohberg
Alumni
Clément Schneider
Gemma Collins
Alexandra Schmidt
AFFILIIERT
Jörg Römbke (enchytraeids, ISO standardisation)
Rüdiger Schmelz (enchytraeids)
Pete Manning (BE-Spring)
Ulrich Burkhardt (Collembolas)
GENOMDATEN
MetaInvert erstellt eine Genomdatenbank für mehrere hundert Arten von wirbellosen Bodenlebewesen: Springschwänze, oribatide Milben, Nematoden, Topfwürmer, Myriapoden und weitere Gruppen. Wir haben bereits Genomsequenzen von über 280 Arten generiert (Abb. 2).
Abb. 2. Taxonomische Beziehungen von über 200 wirbellosen Bodenlebewesen, die im Rahmen des MetaInvert-Projekts genomsequenziert wurden. Der Baum spiegelt den Projektstand vom September 2020 wider. Seitdem sequenzierte Arten (etwa 80) sind nicht enthalten.
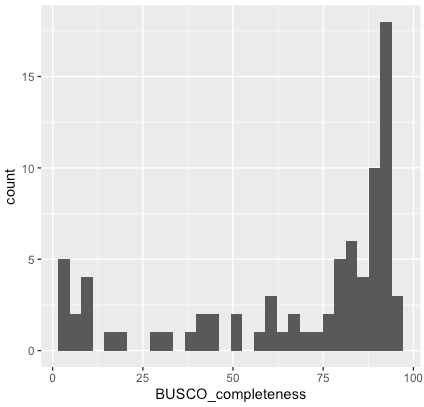
Die Genomdaten in MetaInvert werden meist von einzelnen Individuen generiert. Die meisten dieser Individuen wurden in der Abteilung Bodenzoologie, Senckenberg Museum für Naturkunde Görlitz gesammelt und identifiziert. Die meisten der Genome sind als Illumina short reads sequenziert. Die Genome haben eine Coverage zwischen 2x – 40x und eine unterschiedliche Vollständigkeit (Abb. 3). Wir experimentieren auch mit PacBio Sequel II für Long-Read-Sequenzierungen. Die MetaInvert-Datenbank dient der Verbesserung der Artenidentifikation durch Shotgun-Metagenomik, um evolutionäre Beziehungen, spezielle Anpassungen und Wirt-Mikrobiom-Assoziationen aufzudecken.
Abb. 3. Vollständigkeit der Genome von 80 Bodeninvertebratenarten, die bis zum 1. Mai 2020 sequenziert wurden, geschätzt anhand von BUSCO-Genen.
KOOPERATIONSPROJEKTE
Im Rahmen ihrer Masterarbeit führte Alexandra Schmidt (Goethe-Universität, Frankfurt) bis einschließlich Frühjahr 2021 ein Testprojekt mit künstlich zusammengestellten Artgemeinschaften, sogenannten Mock Communities, durch, um die Genauigkeit und Effizienz des Shotgun-Metagenomik-Ansatzes zu überprüfen. Die Testgemeinschaften wurden aus einer Vielzahl von genomsequenzierten Arten (Oribatiden- und Gamasinenmilben, Nematoden, Springschwänzen, Enchytraeiden, Bärtierchen) in der Abteilung für Bodenzoologie generiert. Mittels dieses Ansatzes wurden Sensitivitäts- und Spezifitätsanalysen bezüglich der taxonomischen Bestimmung und Gewinnung von Biomasseinformationen anhand der Anzahl sequenzierter Reads durchgeführt. Die Ergebnisse zeigen einen statistisch signifikanten Zusammenhang zwischen Biomasse und shotgun-sequenzierter Read-Anzahl. Stärkster Einflussfaktor für die Reads war jedoch die taxonomische Zugehörigkeit. Dies weist auf systemische taxonomische Unterschiede bei der Effizienz der DNA-Extraktion, der Vorbereitungen der Libraries, oder der Sequenzierung hin.
Das Projekt „BE-Spring: Discovering Collembola biodiversity on grasslands with emerging genomic and metagenomic tools“ ist ein von der DFG gefördertes Projekt innerhalb der Biodiversitätsexploratorien. Es läuft von 2020-2023 und wird von Anna Küchler im Rahmen ihrer Doktorarbeit durchgeführt. BE-Spring ergänzt die MetaInvert-Genomdaten mit den Genomen der häufigsten Bodenspringschwänze aus den 150 Exploratories-Grünlandparzellen, um eine Springschwanz-Genom-Referenzdatenbank (SGRD) für Deutschland zu etablieren. BE-Spring implementiert den in MetaInvert etablierten Shotgun-Metagenom-Ansatz, um zu verstehen:
– wie Umweltfaktoren und die Intensivierung der Grünlandnutzung die Zusammensetzung und Funktion von Springschwanzgemeinschaften beeinflussen, und
– wie Springschwänze mit anderen biologischen Komponenten des Grünland-Ökosystems zusammenhängen.
EXPERTISE / METHODEN
- Umwelt-DNA
- Metabarcoding
- Sammlung von Boden-Arthropoden
- Phylogenetik
- Python
- R
- Metagenomik
- Gemeinschaftsökologie
PUBLIKATIONEN
Schneider C, Woehle C, Greve C, et al (2021). Two high-quality de novo genomes from single ethanol- preserved specimens of tiny metazoans (Collembola). GigaScience.
Bálint M, Nowak C, Márton O, et al. (2018). Accuracy, limitations and cost efficiency of eDNA-based community survey in tropical frogs. Molecular Ecology Resources, 18(6), 1415–1426.