Vergleichende Genomik
Evolution und Funktion von Spezies-Interaktionsnetzwerken am Beispiel symbiotisch lebender Pionierarten
Institution: Arbeitskreis für angewandte Bioinformatik, Goethe-Universität, Frankfurt
Symbiotische Gemeinschaften von Organismen sind ein evolutionär sehr erfolgreiches Konzept, um verfügbare Ressourcen in einem Lebensraum optimal zu nutzen. Wir stehen jedoch erst am Anfang, wenn es darum geht, Art und Ausmaß der organismischen Wechselwirkung, ihre molekulare Grundlage sowie die Konsequenzen für die Evolution molekularer Funktionen zu verstehen. In vielen Fällen liegt das an der Komplexität der ökologischen Nische, in die eine symbiotische Gemeinschaft eingebettet ist. Sie beherbergt oft eine Vielzahl von Arten und bietet ein vielfältiges Spektrum an Ressourcen. Eine genaue Abgrenzung der symbiotischen Gemeinschaft und ein mechanistisches Verständnis der Symbiose sowie der daraus resultierenden Synergien ist daher oft schwierig.
In diesem Projekt konzentrieren wir uns auf Pioniergemeinschaften, d.h. auf Metaorganismen, die die Neubesiedelung von Habitaten initiieren. Oft handelt es sich dabei um extreme Lebensräume wie nackte Felsen oder Lavafelder. Auf der Grundlage neu generierter Genomsequenzen der Symbionten annotieren wir die kodierten Protein- Interaktionsnetzwerke und analysieren deren Integration über die symbiotischen Partner. Ein Vergleich mit nah verwandten Arten ohne einen Pionierstatus gibt schließlich Hinweise darauf, über welche evolutionären Wege sich eine Spezies an eine extreme Umwelt angepasst hat.
TBG PROJEKT-TEAM
- Prof. Dr. Ingo Ebersberger (PI)
- Dr. Francisca Segers
- Andreas Blaumeiser
- Julian Dosch
EXPERTISE / METHODEN
- Phylogenomik
- Orthologie-Inferenz
- Genom-Assemblierung
- Vergleichende Genomik
- Mikrobiom-Analyse
- Allgemeine Bioinformatik
- Evolutionsbiologie
- Verhaltensökologie
SEQUENZIERTE GENOME
1 — Lichen hologenom Illumia 120x, PacBio 30x
1 — Plant de novo PacBio 50x, Illumina 50x
De novo genomes: 3
Arten:
Umbilicaria pustulata
Glaux maritima
Ardisia venosa
Lysimachia punctata
Cyclamen purpurascens
PUBLIKATIONEN
The evolutionary making of SARS-CoV-2.
Iruegas R, Dosch J, Sikora et al. (eingereicht). BioRxiv. https://doi.org/10.1101/2021.01.29.428808
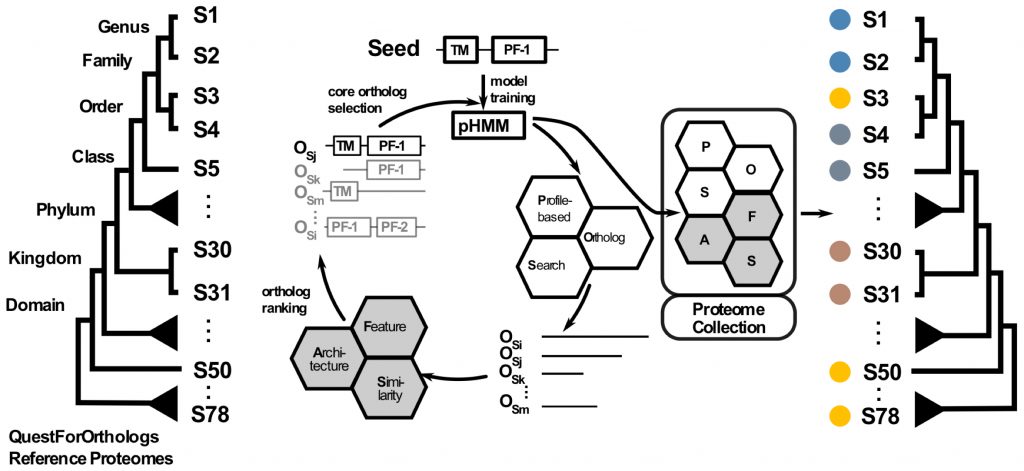
Ten years of collaborative progress in the Quest for Orthologs.
Linard B, Ebersberger I, McGlynn S.E. et al. (2021). Molecular Biology and Evolution, msab098 (online).
Comparative transcriptomics reveal insights into the drought response of the three Panicum species P. bisulcatum, P. laetum and P. turgidum
Kotrade, P, et al.
Plant Gene Volume 23, September 2020, https://doi.org/10.1016/j.plgene.2020.100232
Adjustment of photosynthetic activity to drought and fluctuating light in wheat
Grieco, M. et al. Plant Cell Environ. 2020;1-17; https://doi.org/10.1111/pce.13756
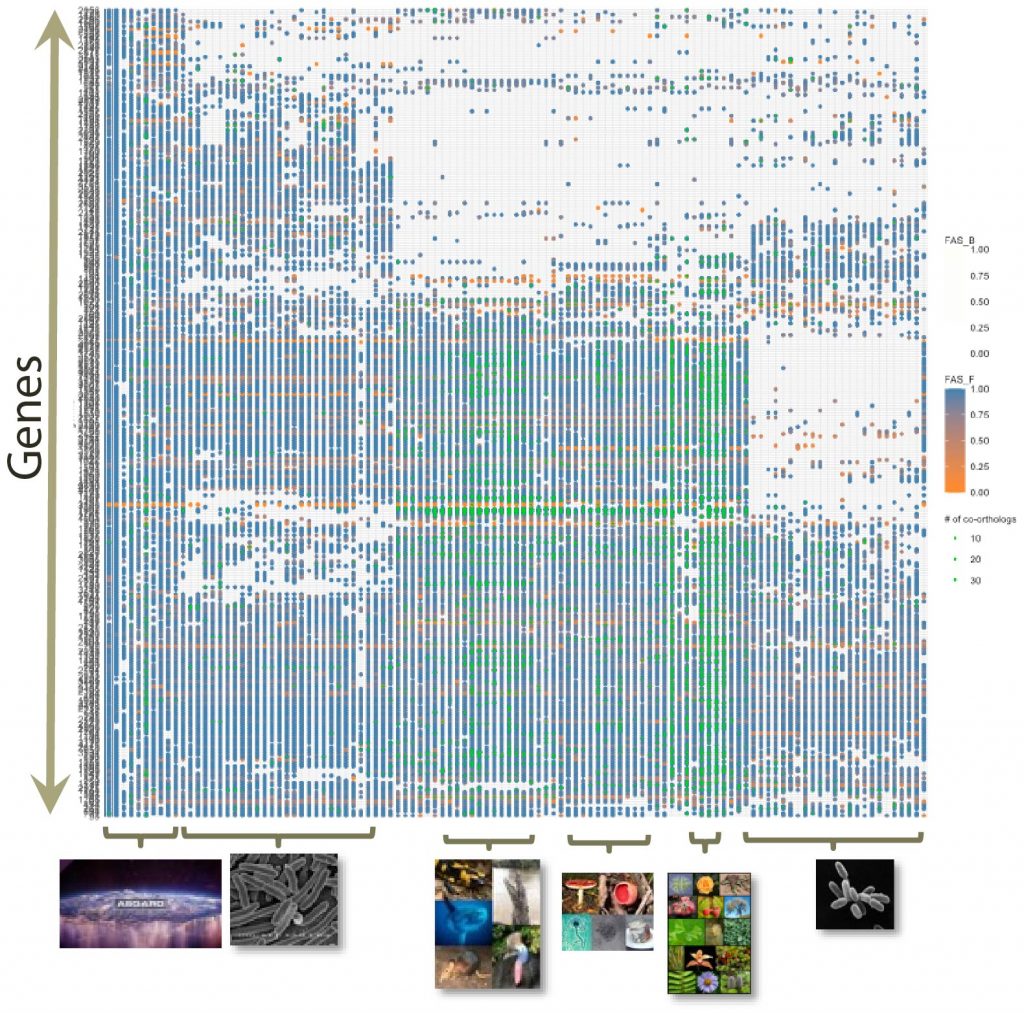
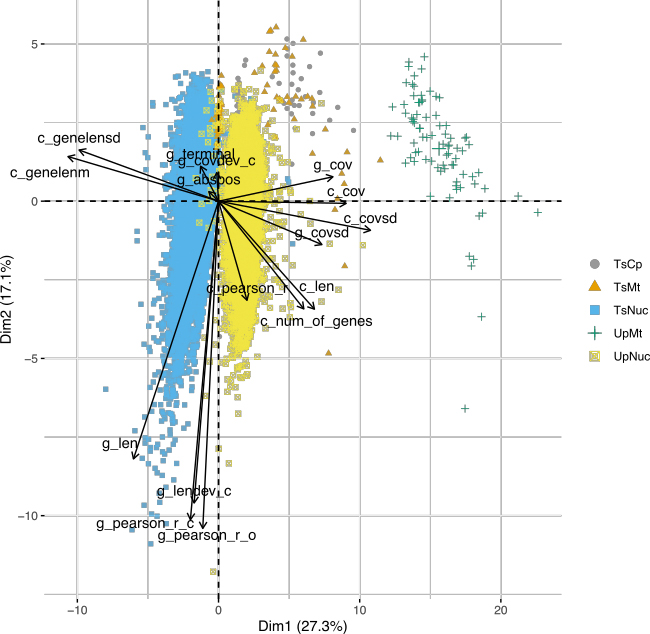
What is in Umbilicaria pustulata? A metagenomic approach to reconstruct the holo-genome of a lichen
Greshake Tzovaras, B.; Segers, F.; Bicker, A.; Dal Grande, F.; Otte, J.; Yahya Anvar, S.; Hankeln, T.; Schmitt, I.; Ebersberger, I.;
Genome Biology and Evolution 2020, eva0049
Advances and Applications in the Quest for Orthologs.
Glover N, et al.
Mol Biol Evol. 2019 Oct 1;36(10):2157-2164. doi: 10.1093/molbev/msz150.#
The evolutionary traceability of a protein
Jain, A. et al.
Genome Biol Evol. 2019 Feb 1;11(2):531-545. doi: 10.1093/gbe/evz008.