Vergleichende Genomik
Evolution von biogenen Funktionselementen und damit verbundenen Verhalten am Beispiel von Seidenspinn- und Köcherbaustrategien bei Köcherfliegen
Foto: Wolfgang Graf
Institution: Senckenberg Gesellschaft für Naturforschung
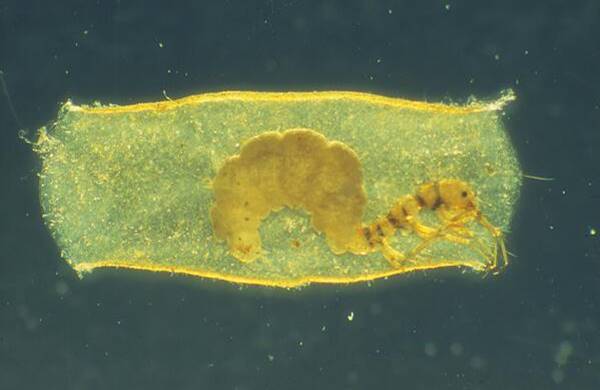
Köcherfliegen, oft als “Unterwasserarchitekten” bezeichnet, haben eine einzigartige Fähigkeit: Sie können Seide unter Wasser spinnen, die dann teilweise in starker Strömung aushärtet. Diese Seide weist adhäsive und faserige Bereiche auf, ist aber zugleich elastisch und hat eine hohe Zugkraft. Köcherfliegen nutzen ihre Seide, um vielfältige Strukturen wie schützende Köcher, Wohnröhren, Fangnetze und Puppengehäuse mit selektiver Ionen-Semipermeabilität zu bauen. Diese Eigenschaften machen die Seide als Konstruktionsmaterial oder für biomedizinische Anwendungen hochinteressant.
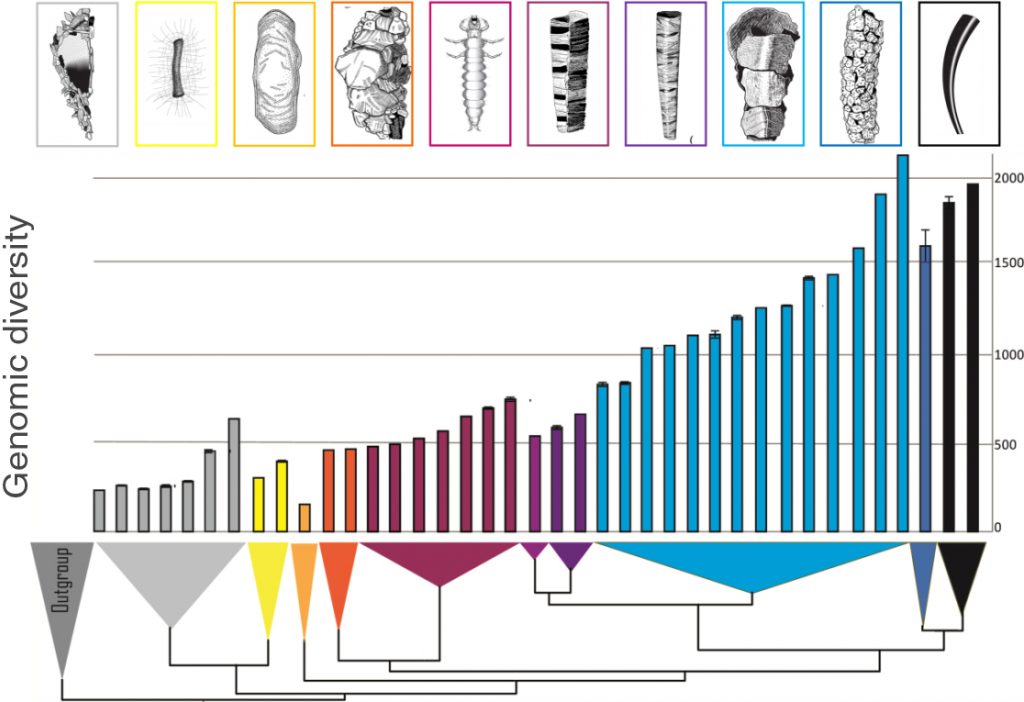
In der Aufbauphase haben wir (1) mehr als 20 Genome verschiedener Köcherfliegenarten sequenziert und assembliert, und entdeckt, dass köcherbildende Arten im Vergleich zu netzbauenden Arten signifikant größere Genome haben, was mit funktionaler Diversifikation und komplexerer Nutzung der Seide zusammenhängen könnte, (2) die Treiber der Genomgrößenvariation identifiziert, (3) strukturelle Unterschiede in den wichtigen L- und H-Fibroin Seiden-Genen charakterisiert und einen Vergleich der Seidengene von Köcherfliegen und deren terrestrischen Schwestergruppe der Lepidoptera unternommen.
In der Entwicklungsphase beschäftigen wir uns mit der Evolution und Charakterisierung von L- und H-Fibroin Genen als zentrale Funktionselemente der Unterwasserseide von Köcherfliegen und anderen Wasserinsekten. Die Verwertbarkeit der Ergebnisse zur rekombinanten Expression von L- und H-Fibroin Genen soll in Kooperation mit Prof. Dr. Philipp Seib und dem Frauenhofer-Institut für Molekularbiologie und Angewandte Ökologie IME untersucht werden.



Gallery Item Name
Lorem ipsum dolor sit amet, consectetur adipisicing elit. Quidem, provident.

Gallery Item Name
Lorem ipsum dolor sit amet, consectetur adipisicing elit. Quidem, provident.
Gallery Item Name
Lorem ipsum dolor sit amet, consectetur adipisicing elit. Quidem, provident.

Gallery Item Name
Lorem ipsum dolor sit amet, consectetur adipisicing elit. Quidem, provident.

Gallery Item Name
Lorem ipsum dolor sit amet, consectetur adipisicing elit. Quidem, provident.
Vergleich des wichtigsten Seidenproteins, h-Fibroin, in verschiedenen Unterordnungen von Köcherfliegen
Die h-Fibroine haben konservierte Termini und Grundmotivstrukturen, weisen jedoch Variation in den sich wiederholenden Modulen sowie im Aminosäurengehalt, hauptsächlich Prolin, auf. Dies könnte mit verschiedenen mechanischen Eigenschaften im Zusammenhang mit der unterschiedlichen Seidenverwendung zusammenhängen und bildet einen Ausgangspunkt für zukünftige Studien zur Untersuchung des Zusammenhangs von Aminosäuremotiven und anderen Sequenzmerkmalen mit quantifizierbaren Seideneigenschaften.
TBG Projekt-Team
- Prof. Dr. Steffen Pauls (PI)
- Jacqueline Heckenhauer, Ph.D.
- Dr. Julio Schneider
- Extern: Prof. Paul Frandsen (Brigham Young University)
Expertise / Methoden
- de novo Genom-Assemblierung und Annotation
- Populationsgenetik
- Phylogenetik
- verankerte Hybridanreicherung



Fotos: Sven Tränkner
Sequenzierte Genome
- 25 de novo Genome verschiedener Köcherfliegenarten (Trichoptera)
PUBLIkationen
Jacqueline Heckenhauer, Russell J. Stewart, Blanca Ríos-Touma, Ashlyn Powell, T. Dorji, Paul B. Frandsen, Steffen U. Pauls (2023). Characterization of the primary structure of the major silk gene, h-fibroin, across caddisfly (Trichoptera) suborders. iScience 26 (8). https://www.sciencedirect.com/science/article/pii/S2589004223013305
Paul B Frandsen, Scott Hotaling, Ashlyn Powell, Jacqueline Heckenhauer, Akito Y Kawahara, Richard H Baker, Cheryl Y Hayashi, Blanca Ríos-Touma, Ralph Holzenthal, Steffen U Pauls, Russell J Stewart (2023). Allelic resolution of insect and spider silk genes reveals hidden genetic diversity, PNAS 120 (18): e2221528120.
Scott Hotaling, Edward R Wilcox, Jacqueline Heckenhauer, Russell J Stewart, Paul B Frandsen (2023). Highly accurate long reads are crucial for realizing the potential of biodiversity genomics, BMC genomics 24 (1):1-9.
Russell J Stewart, Paul B Frandsen, Steffen U Pauls, Jacqueline Heckenhauer (2022). Conservation of three-dimensional structure of Lepidoptera and Trichoptera L-Fibroins for 290 million years, Molecules 27 (18): 5945.
Jacqueline Heckenhauer, Paul B Frandsen, John S Sproul, Zheng Li, Juraj Paule, Amanda M Larracuente, Peter J Maughan, Michael S Barker, Julio V Schneider, Russell J Stewart, Steffen U Pauls (2022). Genome size evolution in the diverse insect order Trichoptera, GigaScience 11: giac011, https://doi.org/10.1093/gigascience/giac011.
Blanca Ríos-Touma, Ralph W Holzenthal, Ernesto Rázuri-Gonzales, Jacqueline Heckenhauer, Steffen U Pauls, Caroline G Storer, Paul B Frandsen (2022). De novo genome assembly and annotation of an Andean caddisfly, Atopsyche davidsoni Sykora, 1991, a model for genome research of high-elevation adaptations, Genome Biology and Evolution 14 (1): evab286, https://doi.org/10.1093/gbe/evab286.
Xuankun Li, Emily Ellis, David Plotkin, Yume Imada, Masaya Yago, Jacqueline Heckenhauer, Timothy P Cleland, Rebecca B Dikow, Torsten Dikow, Caroline G Storer, Akito Y Kawahara, Paul B Frandsen (2021): First annotated genome of a mandibulate moth, Neomicropteryx cornuta, generated using PacBio HiFi sequencing, Genome Biology and Evolution 13 (10): evab229, https://doi.org/10.1093/gbe/evab229.
Scott Hotaling, John S Sproul, Jacqueline Heckenhauer, Ashlyn Powell, Amanda M Larracuente, Steffen U Pauls, Joanna L Kelley, Paul B Frandsen (2021). Long reads are revolutionizing 20 years of insect genome sequencing, Genome Biology and Evolution 13 (8): evab138, https://doi.org/10.1093/gbe/evab138.
Lindsey K Olsen*, Jacqueline Heckenhauer*, John S Sproul, Rebecca B Dikow, Vanessa L Gonzalez, Matthew P Kweskin, Adam M Taylor, Seth B Wilson, Russell J Stewart, Xin Zhou, Ralph Holzenthal, Steffen U Pauls, Paul B Frandsen (2021). Draft genome assemblies and annotations of Agrypnia vestita Walker, and Hesperophylax magnus Banks reveal substantial repetitive element expansion in tube case-making Caddisflies (Insecta: Trichoptera). Genome Biology and Evolution, 13(3), evab013.
Jacqueline Heckenhauer, Steffen U. Pauls (2021). Faszinierende Unterwasserarchitekten. Natur Forschung Museum 151: 74-76.
Jacqueline Heckenhauer, Paul B Frandsen, Deepak K Gupta, Juraj Paule, Stefan Prost, Tilman Schell, Julio V Schneider, Russell J Stewart, Steffen U Pauls (2019). Annotated draft genomes of two caddisfly species Plectrocnemia conspersa CURTIS and Hydropsyche tenuis NAVAS (Insecta:Trichoptera). Genome Biology and Evolution 11 (12): 3445-3451. https://doi.org/10.1093/gbe/evz264