Vergleichende Genomik
Strategien zur Optimierung der Analyse von Genfluss bei der Artbildung am Beispiel von Giraffen, Seehunden und Robben
Institution: Senckenberg Gesellschaft für Naturforschung
kurzbeschreibung
Genfluss zwischen Arten widerspricht dem biologischen Artkonzept, das Arten als reproduktiv isolierte Einheiten identifiziert. Wie unsere Analysen zeigten, findet man aber klare Evidenz für Genfluss z.B. in Bären [4] und Walen (Árnason et al. 2018). Damit stellt sich die Frage, was eine Art ausmacht, wenn Genfluss häufiger als erwartet vorkommt. Dies lässt sowohl für die Artdefinition und -bildung, als auch den Austausch von genetischer Variabilität und damit Anpassungspotenzial (u.a. Umweltveränderungen, Krankheiten) eine große Bedeutung vermuten. Genflussanalysen sind allerdings zurzeit aufwändig und wenig standardisiert, was trotz des hohen Potenzials für die Grundlagenforschung die allgemeine Anwendung erschwert. Das langfristige Ziel des Projektes ist es, Genfluss in die Artdefinition mit einzubeziehen, sowie im Bereich Conservation Genetics zur Artabgrenzung zu nutzen, um Biodiversität auf genomischer Ebene besser zu verstehen und schützen zu können.
Zur umfassenden Analyse von Genfluss in Wirbeltieren wurden in diesem Projekt für zehn Seehundarten neue Genome sequenziert und assembliert. Außerdem wurden Resequen zierungsdaten von 88 Bären (77 Braunbären, 10 Eisbären, ein Hybrid), 54 Finnwalen, 173 Giraffen und im Rahmen des TBG-Exchange-Programms (Dr. Ferrette) 65 Fächerfischen generiert. Umfassende Analysen der Giraffengenome zeigten, dass es bei dieser Gruppe zwar keinen Genfluss gibt, dass sich Giraffen jedoch in vier klar getrennte Arten mit insgesamt sieben Unterarten aufteilen, die seit mehr als 200.000 Jahren getrennt voneinander evolvieren (Coimbra et al. 2021). Diese und weitere geplante Arbeiten tragen unmittelbar zu Artenschutz Entscheidungen der IUCN Okapia and Giraffe Specialist Group, deren Mitglied Janke seit 2016 ist, bei. Die Veröffentlichungen der Analysen der Bären-, Wal-, und Fischgenome werden in 2021 erfolgen. Weiterhin wurden erste Schritte unternommen, um die Methodik zur Analyse von Genfluss besser nutzbar und standardisiert zu machen.
Highlights
Migrationsraten unter vier verschiedenen Giraffengruppen
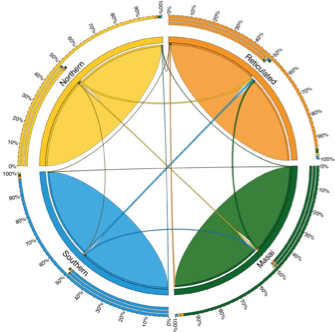

Kreisdiagramm mit aktuellen Migrationsraten unter den vier Giraffengruppen, geschätzt von BayesAss und dargestellt mit Bändern, die eine Art mit einer anderen verbinden. Die Farbkodierung bezeichnet die vier Arten.
Genfluss-Signale für Bartenwale:

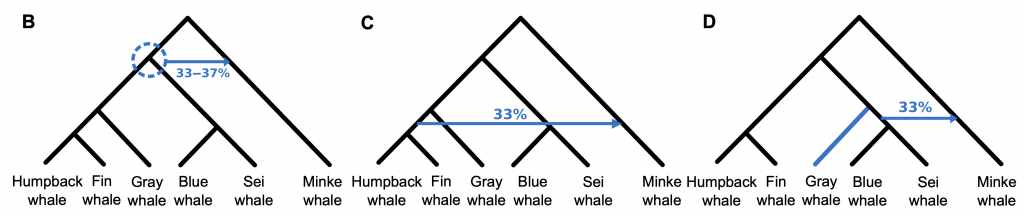
Genfluss-Signale für Bartenwale, abgeleitet von PhyloNet. (Wurzel-Netzwerke basierend auf 34.192 20-kbp-Genomfragmenten). Die Vernetzungen sind durch blaue Pfeile dargestellt, die Vererbungswahrscheinlichkeit ist darüber oder darunter angegeben.
Projektteilnehmer
- Prof. Dr. Axel Janke (PI)
- Dr. Menno de Jong
- Dr. Bruno Ferrette
- MSc Sven Winter
- MSc Raphael Coimbra
- MSc Magnus Wolf
- MSc Marcel Nebenfuhr
- MSc David Prochotta
Sequenzierte Genome
450 — Giraffen Illumina 15x
6 — Furchenwale de novo PacBio and 10XGenomics
50 — Finwale Illumina 20x
250 – Braunbären Illumina 20x
Expertise / Methoden
Genomassemblierung
Analyse evolutionärer Netzwerke
Genfluss D-Stat und Derivate
PUBLIKATIONEN
Coimbra et al „Whole-genome analysis of giraffe supports four distinct species“. Current Biology, Vol. 31, Issue 13,12 July 2021
Stefan Prost, Sven Winter, Jordi De Raad, Raphael T F Coimbra, Magnus Wolf, Maria A Nilsson, Malte Petersen, Deepak K Gupta, Tilman Schell, Fritjof Lammers, Axel Janke, Education in the genomics era: Generating high-quality genome assemblies in university courses, GigaScience, Volume 9, Issue 6, June 2020,
Sven Winter, Stefan Prost, Jordi De Raad, Raphael T. F. Coimbra, Magnus Wolf (…) Maria A. Nilsson, Axel Janke, Chromosome-level genome assembly of a benthic associated Syngnathiformes species: the common dragonet, Callionymus lyra,Gigabyte,1,2020
Árnason, Úlfur, et al. “Whole-genome sequencing of the blue whale and other rorquals finds signatures for introgressive gene flow.” Science advances 4.4 (2018)
TBG GENOMSTATISTIK:
| De novo genomes | 3 |
| Re- sequenced genomes | 6 |
| Total genomes: | 9 |
| Total data: | 0,751 Tera bases |
Analysierte arten:
| Arten: | Datum | NCBI | Coverage Quality (planned) | |
| Giraffen: | 01.01.18 | Yes | 15x | re-sequenziert |
| Nord-Giraffe (Giraffa camelopardalis) Nubian | 01.01.19 | No | (15x) | re-sequenziert |
| Nord-Giraffe (G. camelopardalis) Rothschild’s | 01.01.19 | No | (15x) | re-sequenziert |
| Nord-Giraffe (G. camelopardalis) West African | 01.01.19 | No | (15x) | re-sequenziert |
| Netz-Giraffe (G. reticulata) | 01.01.19 | No | (15x) | re-sequenziert |
| Masai-Giraffe (Gi. tippelskirchi) Masai | 01.01.19 | No | (15x) | re-sequenziert |
| Masai-Giraffe (G. tippelskirchi) Luanga Valle NP | 01.01.19 | No | (15x) | re-sequenziert |
| Southern giraffe (G. giraffa) Angolan | 01.01.19 | No | (100x) | de-novo (gold) |
| Süd-Giraffe (G.a giraffa) South African | 01.01.19 | No | (15x) | re-sequenziert |
| Bären: | ||||
| Syrischer Braunbär (Ursus arctos syriacus) male | 01.07.18 | No | (20x) | re-sequenziert |
| Syrischer Braunbär (Ursus arctos syriacus) female | 01.07.18 | No | (20x) | re-sequenziert |
| Kamchatka-Braunbär (Ursus arctos beringianus) | 01.07.18 | No | (20x) | re-sequenziert |
| Wale: | ||||
| Atlantischer Nordkaper (Eubalena glacialis) | 06.04.18 | Yes | (10x) | re-sequenziert |
| Seiwal (Balaenoptera borealis) | 06.04.18 | Yes | (10x) | re-sequenziert |
| Seiwal (Balaenoptera borealis) | 06.04.18 | Yes | (10x) | re-sequenziert |
| Blauwal (Balaenoptera musculus) | 06.04.18 | Yes | (30x) | re-sequenziert |
| Buckewal(Megaptera novaeangliae) | 06.04.18 | Yes | (10x) | re-sequenziert |
| Finnwal (Balaenoptera physalus) | 06.04.18 | Yes | (100x) | de-novo (gold) |
| Minkwal (Balaenoptera acutorostrata) | 06.04.18 | Yes | (10x) | re-sequenziert |
| Grauwal (Eschrichtius robustus) | 06.04.18 | Yes | (10x) | re-sequenziert |
| Grauwal (Eschrichtius robustus) | 06.04.18 | Yes | (10x) | re-sequenziert |
| Hippopotamus (Hippopotamus amphibius) | 06.04.18 | Yes | (60x) | de-novo gold |